【机构】 福建省农业科学院食用菌研究所;
【摘要】 利用SRAP分子标记技术对福建省农科院食用菌研究所保藏的32个茯苓菌株进行DNA指纹图谱的扩增,通过10对引物获得23条特异性条带,并用NTSYSpc-2.02j软件进行聚类分析结果,供试的32个菌株在0.38相似值分为两类,其中菌株P6283单独聚为一类,其余的31个菌株聚为一类。这31个菌株在0.76的相似值又可以分为3个亚类群,其中有28个菌株可通过该组标记予以区分。供试的32个茯苓菌株,除P6283外,虽然源自不同地区,但遗传相似系数较高,表明所保藏的茯苓菌株整体遗传背景较窄。
【关键词】 茯苓菌株; SRAP技术; DNA指纹分析;
【文内图片】

2个茯苓菌株的SRAP扩增图谱(引物对me1-em7)
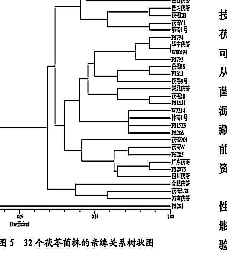
32个获答菌株的亲缘关系树状图
【基金】 福建省发改委“五新”推广项目“茯苓松蔸标准化栽培技术研究与示范”[编号:闽发改农业(2010)452号];福建省科技厅公益类项目“茯苓GAP关键栽培技术研究”(编号:2010R1022-2)的部分研究内容
【分类号】S567.32





