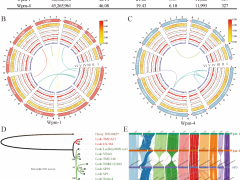
羊肚菌科(Morchellaceae)包括可食用的羊肚菌和块菌,以及其他不可食用的种类。羊肚菌被普遍认为是腐生真菌,但由于缺乏系统的基因组比较和进化分析,学界对其能否与植物形成生物营养关系存在长期争议,且对其营养模式演化过程尚不明晰。
近日,中国科学院西北生态环境资源研究院联合美国密歇根州立大学、日本冲绳科学技术大学院大学、美国劳伦斯伯克利国家实验室、美国能源部联合基因组研究所、法国国家农业食品与环境研究院(INRAE),对覆盖全球羊肚菌主要谱系的高质量基因组数据进行了深入分析,揭示了羊肚菌科腐生菌的基因组基础,以及羊肚菌科块菌进化为依赖于植物宿主营养的外生菌根生活方式的基因组特征。
研究表明,羊肚菌保留了完整的植物降解酶基因组库,具备典型的腐生能力。相比之下,羊肚菌科的块菌,如Leucangium spp.显示出转座元件富集、降解酶减少等与外生菌根共生真菌一致的基因组特征。同时,研究发现羊肚菌主要是MAT1-1或MAT1-2异宗结合,但也有少数羊肚菌科物种是同宗结合。进一步,研究指出羊肚菌或处于“腐-共生”连续体的中间区域。
该研究弥补了关于羊肚菌腐生能力及其与植物潜在生物营养关系长期存在的认识空白,为学界理解土壤真菌营养模式的复杂演化提供了重要基因组证据。
相关研究成果以Pangeneric analyses reveal the divergent genome evolution and ecologies between morels and truffles in the Morchellaceae为题,发表在《当代生物学》(Current Biology)上。研究工作得到国家自然科学基金委员会等的支持。
原文链接:https://doi.org/10.1016/j.cub.2025.07.037